 |
Biologie-Kurs: Molekulargenetik |
|
|---|---|---|
| Zurück zum ersten Teil |
![]() Überlege dir, wie viele Schriftzeichen in unserer Sprache verwandt
werden.
Überlege dir, wie viele Schriftzeichen in unserer Sprache verwandt
werden.
![]() Welches Nachrichtensystem, Nachrichtentechnik oder Informationssystem kennst
du, das mit weniger Zeichen auskommt ?
Welches Nachrichtensystem, Nachrichtentechnik oder Informationssystem kennst
du, das mit weniger Zeichen auskommt ?
Bei der DNS stehen uns mit den vier verschiedenen Nukleotiden sogar
mehr Zeichen zur Verfügung als beim Morsen. Informationsübertragung
und Informationsspeicherung ist demnach grundsätzlich mit der DNS
möglich. Die Information ist bei der DNS in der unterschiedlichen
Sequenz der vier verschiedenen Nukleotide gespeichert. Dabei ist die Information
in einem DNS-Doppelstrang eigentlich doppelt vorhanden: einmal in der Nukleotidsequenz
des einen Einzel-Stranges und dann "spiegelbildlich" in der Sequenz des
komplementären Einzel-Stranges. Die Information liegt praktisch als
"Negativ" und als "Positiv" vor. Dies ist zudem eine sehr sichere Informationsspeicherung.
Denn gehen aus einer Stranghälfte Nukleotide verloren, können
nur die zum anderen Strang komplementären Nukleotide wieder eingebaut
werden. (Von einem Negativ kann man Abzüge herstellen: Positive. Und
bei einem verlorenen Negativ kann man vom Papierabzug auch wieder ein Negativ
herstellen.)
| a) Konservatives Replikationsmodell:
Man geht davon aus, dass nach der Replikation neben dem alten DNS-Doppelstrang ein kompletter neuer DNS-Doppelstrang aufgebaut wurde. |

konservatives Replikationsmodell verändert nach Freeman |
| b) Dispersives Replikationsmodell
Nach dem dispersiven Replikationsmodell bekommt man neue DNS-Doppelstränge, die abwechselnd aus alten und neuen DNS-Doppelstrang-Stücken zusammengesetzt sind.
|

dispersives Replikationsmodell verändert nach Freeman |

(hdm) semikonservatives Trickmodell |

semikonservatives Replikationsmodell verändert nach Freeman |
Die Klärung, welches dieser Modelle bei der DNS-Replikation verwirklicht wurde, lieferten die Experimente von Meselson und Stahl (1958).
Kontrollen für das Experiment von Meselson und Stahl:

Bakterien werden in einem Medium mit schweren Stickstoff 15N gezogen |

Bakterien werden in ein Medium mit normalem Stickstoff 14N überführt |

Von beiden Kulturen werden zur Kontrolle Proben entnommen, die Bakterien zerstört und im Dichtegradienten zentrifugiert |
Zentrifugenglas
|
Zentrifugenglas
|
Überlege, wie das Ergebnis nach einem Replikationszyklus
![]() Und so sah das Ergebnis nach einem Replikationszyklus
aus:
Und so sah das Ergebnis nach einem Replikationszyklus
aus:
... und damit war noch nicht geklärt, nach welchem Modell die
Replikation abläuft.
Überlege deshalb, wie das Versuchsergebnis nach zwei Replikationszyklen
| Und so sah das Ergebnis des Meselson-Stahl-Versuchs nach zwei Replikationszyklen tatsächlich aus. |
Allerdings stellte sich die Replikation doch etwas komplizierter dar, als das zunächst sehr einfache Modell erwarten ließ.

(hdm) Simulation der Relikation |
Unterschiede zwischen DNS und RNS:

(hdm) Ribose-Desoxyribose |

Nukleotid mit Thymin |
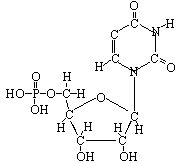
Nukleotid mit Uracil |

(hdm) Transkription im Trick |

(hdm) Simulation der Translation |

(hdm) t-RNS in der Kleeblattstruktur |
(hdm) t-RNS mit einer Aminosäure |
(hdm) t-RNS mit Anti-Codon |
| die wichtigen Teile einer transfer-RNS | ||

(hdm) Translation mit Faltung des Proteins im Trick |
Vorüberlegungen:
Kann rein rechnerisch ein Nukleotid-Paar eine Aminosäure codieren?
| AA | AC | AG | AU |
| CA | CC | CG | CU |
| GA | GC | GG | GU |
| UA | UC | UG | UU |
Mögliche Paare:
Bei 4 Nukleotiden auf 2 Stellen verteilt ergeben sich 42
= 16 Möglichkeiten.
16 mögliche Paare reichen nicht aus, um 20 verschiedene Aminosäuren
eindeutig zuzuordnen.
Kann rein rechnerisch ein Nukleotid-Triplet eine Aminosäure
codieren?
Mögliche Triplets:
AAA, AAC, ....usw.
Insgesamt ergeben sich bei 4 Nukleotiden verteilt auf 3 Stellen 43
= 64 Möglichkeiten.
Mit 64 Nukleotid-Kombinationen lassen sich 20 Aminosäuren
eindeutig zuordnen. Allerdings haben wir drei mal soviel Möglichkeiten
wie notwendig sind.
Kann rein rechnerisch ein Nukleotid-Quardruplet eine Aminosäure
codieren?
Mögliche Quadruplets:
AAAA, AAAC, ....usw.
Insgesamt ergeben sich bei 4 Nukleotiden verteilt auf 4 Stellen 44
= 256 Möglichkeiten.
Mit 256 Nukleotid-Kombinationen lassen sich 20 Aminosäuren
eindeutig zuordnen. Allerdings haben wir mehr als zehn mal soviel Möglichkeiten
wie notwendig sind.
Aus den Vorüberlegungen wird klar, dass mindestens drei Nukleotide für die Codierung einer Aminosäure notwendig sind. Welche Lösung wurde aber in der Natur verwirklicht?
Gedanken-Experimente zum Knacken des Codes:
(Es sind waren selbstverständlich noch viele andere
Codierungsmöglichkeiten denkbar. Wir beschränken uns hier aber
auf die Untersuchung von 3er- und 4er-Rastern.)
Inzwischen (1961, Nirenberg und Matthaei) war es möglich eine
künstliche m-RNS z.B. Poly-Uracil herzustellen
----UUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUU---
und in einem zellfreien System in vitro damit Polypeptide zu produzieren.
(Ein zellfreies System erhält man z.B. indem man E.Coli Bakterien
aufbricht und zentrifugiert und den zellfreien Überstand benutzt.
Diesem werden noch gereinigte E.Coli-Ribosomen hinzugefügt.)
Das identifizierte Protein bestand nur aus der Aminosäure Phenylalanin:
![]() Können wir damit entscheiden, ob es sich um
einen Triplet- oder Quadruplet-Code handelt?
Können wir damit entscheiden, ob es sich um
einen Triplet- oder Quadruplet-Code handelt?
![]() Was können wir trotzdem für später festhalten?
Was können wir trotzdem für später festhalten?
Vielleicht hilft das folgende Poly-UG als m-RNS bei der Entscheidung.
..UGUGUGUGUGUGUGUGUGUGUGUGUGUGUGUG
![]() Was für Polypeptid wäre nach einem Triplet-Raster
zu erwarten?
Was für Polypeptid wäre nach einem Triplet-Raster
zu erwarten?
![]() Was für Polypeptid wäre nach einem Quadruplet-Raster
zu erwarten?
Was für Polypeptid wäre nach einem Quadruplet-Raster
zu erwarten?
Im Experiment wurde ein Polypeptid aus den beiden Aminosäuren
Valin und Cystein ermittelt:
- Cys- Val - Cys - Val - Cys - Val -Cys - Val
- Cys - Val -
![]() Welchen Schluss kann man aus diesem Versuchsergebnis ziehen?
Welchen Schluss kann man aus diesem Versuchsergebnis ziehen?
Durch eine Vielzahl solcher Versuche bekam man Basenkombinationen, die man einzelnen Aminosäuren zuordnen konnte. Der eigentliche Durchbruch gelang aber erst Khorana , der im zellfreien System Basentriplets anbot, die auch an t-RNS gebunden wurden. Mit Hilfe von radioaktiver Markierung konnten damit die verschiedenen Triplets den verschiedenen Aminosäuren zugeordnet werden und die Zuordnung in einer Tabelle dargestellt werden:
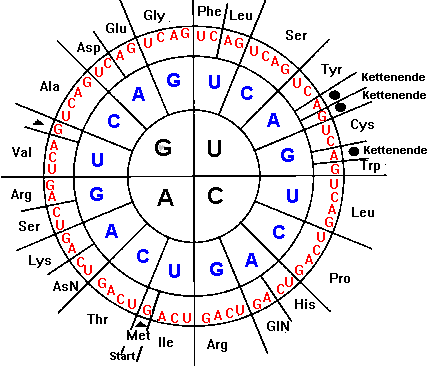
verändert nach Abitools |
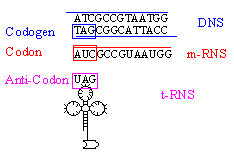 Hier sind noch ein paar Begriffe
zu klären:
Hier sind noch ein paar Begriffe
zu klären:
Ein Triplet auf der m-RNS, das eine Aminosäure codiert und dessen
Informationsgehalt in der Codesonne abgelesen werden kann, heißt:
Codon.
Das zu diesem Codon komplementäre Triplet auf der DNS, von dem
also die m-RNS abgelesen wurde, heißt: Codogen.
Das Basentriplet an der t-RNS, das komplementär zum Codon ist,
heißt: Anticodon.
![]() Welche Aminosäuresequenz würde man im zellfreien System für
die folgende m-RNS bekommen?
Welche Aminosäuresequenz würde man im zellfreien System für
die folgende m-RNS bekommen?
GUGACGCCCGGGAAAUUUAGCGCAGACUAUUCUUGUUCGUGC
(Wir lesen von links nach rechts und beginnen mit der
ersten Base).
Beim Arbeiten mit der Codesonne sind dir sicher einige Dinge aufgefallen:
Deshalb hier nochmals die Codesonne, damit du nicht zu weit blättern
musst.

verändert nach Abitools |
 ausdrucken, damit du nicht immer zurückblättern
musst.
ausdrucken, damit du nicht immer zurückblättern
musst.
Übungen zum genetischen Code:
![]() Hier sollst du Transkription und Translation "in vivo" durchführen.
Hier sollst du Transkription und Translation "in vivo" durchführen.
Du sollst also zur abgebildeten DNS einen M-RNS-Strang komplementär
zum oberen Einzelstrang ablesen und anschließend (von
links beginnend) bei einem Start-Codon mit der Translation beginnen,
bis bei einem Stop-Codon die Translation abbricht.

Undhier
Einbau falscher Basen: Aber Fehler entstehen immer wieder und
diese Fehler sind auch vorprogrammiert. Abweichend vom bisher Gelernten
können nämlich durchaus bei der Replikation oder Transkription
den eigentlichen Nukleotiden analoge Nukleotide eingebaut werden wie z.B.
Bromuracil.
Bromuracil kommt normal in der Keto-Form vor. Hin und wieder lagert
es sich jedoch für kurze Zeit in die Enol-Form um.
Damit kann also ein Basen-Paar A-T durch ein Paar G-C ausgetauscht
werden
Damit kann umgekehrt das Paar Cytosin-Guanin dirch Thymin-Adenin
ersetzt werden.
Wie sich ein derartiger Austausch auswirken kann, wollen wir mit den
folgenden Beispielen durchspielen:
Wir beziehen uns auf ein Stück der DNS von der Länge eines
Tripletts. Salpetrige Säure wirkt ein und verändert das Cytosin
in der Mitte zu Uracil. Nach zwei Replikationen lesen wir bei einem unveränderten
ursprünglichen Strang und einem mutierten Strang (vom oberen Einzelstrang)
die m-RNS ab.
Diesen beiden Beispiele für Mutationen durch salpetrige Säure
zeigen die beiden Extremergebnisse von Punktmutationen: Entweder wirkt
sich die Mutation gar nicht aus, weil trotzdem die gleiche Aminosäure
eingebaut wird, oder es gibt wegen dem Stop-Codon überhaupt kein Protein
mehr. Dazwischen sind sämtliche Abstufungen möglich. Das Protein
könnte durch den Einbau einer anderen Aminosäure nicht, leicht
oder stark verändert sein. Z.B. könnte eine einzige andere Aminosäure
dafür sorgen, dass ein Enzym nicht mehr funktioniert,
weil das aktive Zentrum dadurch betroffen ist.
Rastermutationen (ebenfalls Punktmutationen) sind genauso kleinste
Änderungen der Erbinformation, die jedoch fatale Folgen verursachen
können. Sie können z.B. durch Acridin-Farbstoffe verursacht werden,
die z.B. bei der Replikation anstelle einer Base eingebaut werden und dann
jederzeit wieder verloren gehen können. Nach der nächsten Replikation
ist der DNS Doppelstrang um eine Base kürzer.
Gegenüber dem oberen Satz ist jetzt eine kleine Änderung eingetreten:
Keine großen Probleme gibt es jedoch, wenn gleich 3 Nukleotide
verschwinden
oder dazu kommen:
Hier folgt eine Tabelle mit Beispielen für Mutagene:
Pyrimidin-Dimere können entstehen, wenn in der DNS z.B. zwei Thymin-Basen
nebeneinander stehen und statt mit dem komplimentären Adenin auf der
anderen Seite des Stranges untereinander Wasserstoffbrückenbindungen
eingehen.
 Beispiel 1,
Beispiel 1,  Beispiel 2,
Beispiel 2,  Beispiel 3.
Beispiel 3.
4) Die Fähigkeit zur Mutabilität
Punktmutationen: Die hier besprochenen Mutationen stellen die kleinstmöglichen
Änderungen der Erbinformation dar.
Mutation bedeutet eine sprunghafte Änderung der Erbinformation. Im
Gegensatz dazu hatten wir bisher gesehen, dass peinlich genau auf eine
exakte Verdoppelung und Weitergabe der Erbinformation Wert gelegt wurde.

(hdm) Bromuracil ![]() Überlege,
Überlege,
1) ob Bromuracil eine Purin- oder Pyrimidinbase ist,
2) welcher Base (A, C, G oder T) das Paarungsverhalten von Bromuracil
in der Keto-Form entspricht und
3) mit welcher komplementären Base sich Bromuracil (Keton-Form)
paaren wird.
![]() Überlege,
Überlege,
1) welcher Base (A, C, G oder T) das Paarungsverhalten von Bromuracil
in der Enol-Form entspricht und
2) mit welcher komplementären Base sich Bromuracil (Enol-Form)
paaren wird.
Einwirkung von Säuren: Die Zugabe von schwacher salpetriger
Säure kann zu der in der Abbildung sichtbaren Veränderung führen:
Cytosin wird zu Uracil
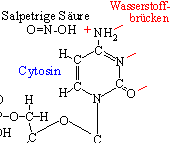
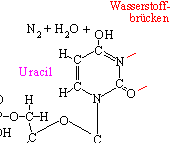
![]() Überlege,
welches Basen-Paar in diesem Fall durch die salpetrige Säure ersetzt
wird.
Überlege,
welches Basen-Paar in diesem Fall durch die salpetrige Säure ersetzt
wird.

(hdm) Mutation durch salpetrige Säure![]() Bestimme mit Hilfe der Codesonne, welche Aminosäure ursprünglich
eingebaut werden sollte und welche jetzt in das Polypeptid eingebaut wird.
Bestimme mit Hilfe der Codesonne, welche Aminosäure ursprünglich
eingebaut werden sollte und welche jetzt in das Polypeptid eingebaut wird.
![]() Welche Folgen kann diese Veränderung für die Funktion des zu
bildenden Polypeptids haben?
Welche Folgen kann diese Veränderung für die Funktion des zu
bildenden Polypeptids haben?
Das nebenstehende Schaubild ist noch unvollständig.

(hdm) Mutation durch salpetrige Säure ![]() Bestimme die m-RNS-Abschnitte, wenn vom oberen Strang abgelesen wird.
Bestimme die m-RNS-Abschnitte, wenn vom oberen Strang abgelesen wird.
![]() Welche Aminosäuren codieren diese beiden Codons?
Welche Aminosäuren codieren diese beiden Codons?
![]() Wie kann man das vorangehende Ergebnis erklären?
Wie kann man das vorangehende Ergebnis erklären?
Schauen wir uns das folgende Beispiel an:
links steht ein Satz in kommafreier Schreibweise wie bei der DNS, rechts
ist der Inhalt durch das Triplet-Raster sichtbar gemacht.
--EINGENISTAUSDNS--

--EINSGENISTAUSDN--
![]() Was ist hier passiert?
Was ist hier passiert?
![]() Wie ist der Inhalt zu verstehen und welche Konsequenzen wird dies haben?
Wie ist der Inhalt zu verstehen und welche Konsequenzen wird dies haben?
verschwinden:
--EINGENAUSDNS--

--EINGEN?ß@ISTAUSDNS--

![]() Um was für eine Mutation würde es sich beim folgenden Beispiel
handeln?
Um was für eine Mutation würde es sich beim folgenden Beispiel
handeln?
--EINRENISTAUSDNS--
Mutagen
Wirkungsweise
Chemische Agenzien
.
Basenanaloga
Sie werden anstelle der richtigen Basen in die DNS eingebaut, paaren
sich anders als die ursprünglichen Basen
Basen-verändernde Verbindungen
Chemische Substanzen, die Veränderungen an den Basen hervorrufen,
so dass diese danach anders paaren
Strahlen
.
Ionisierende Strahlen
bewirken DNS-Strangbrüche
UV-Licht
Bildung von Pyrimidin-Dimeren
Zur Reduzierung von Mutationen gibt es jedoch auch Reparatur-Mechanismen,
die am Beispiel der Pyrimidin-Dimere recht gut untersucht sind.
Quiz 2.Teil
Gesamt-Quiz
Die Ein-Gen-Ein-Enzym-Hypothese,
die jetzt folgen sollte, schauen wir uns beim Kollegen Bobeth an: Genwirkkette.
Ein Online-Kurs ist bei den hohen Telefongebühren in Deutschland
zu teuer??
Dann eben als Offline-Kurs
Gegen eine geringe Bearbeitungsgebühr können Sie sich diese
Zeit sparen und die neueste Version des interaktiven Lernprogramms zur
Offline-Bearbeitung bekommen. Wie?
Verbesserungsvorschläge und Kommentare an Hans-Dieter
Mallig (hans-dieter@mallig.de)


